Ein weiteres Papier des SFB1551 ist erschienen! "Abstimmung der Funktionalität von Designer-Translationsorganellen mit orthogonalen tRNA-Synthetase/tRNA-Paaren" wurde im Journal of Molecular Biology veröffentlicht.
Abstrakt
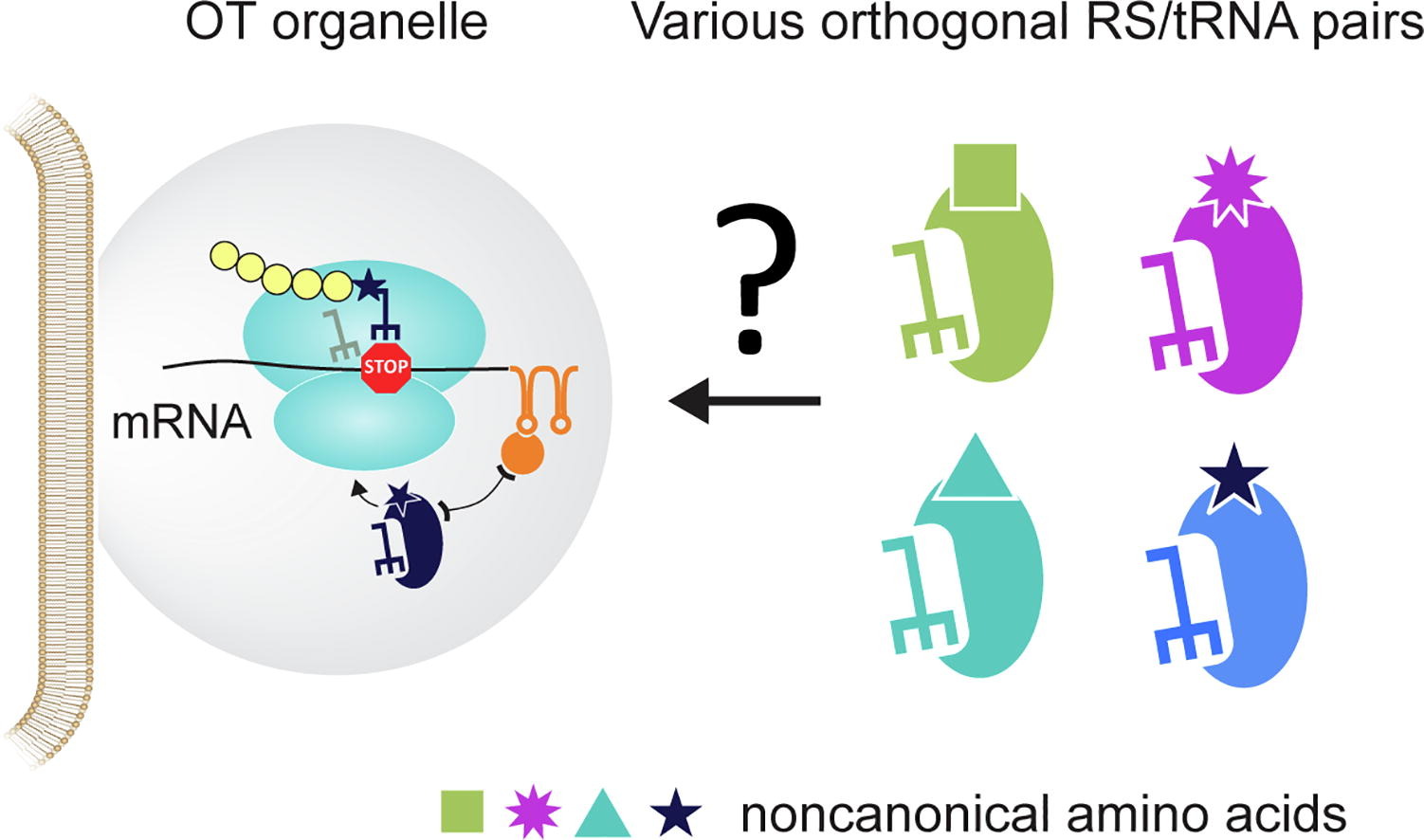
Der ortsspezifische Einbau von nicht-kanonischen Aminosäuren (ncAAs) kann durch die Technologie der genetischen Code-Expansion (GCE) realisiert werden. Es wurden verschiedene orthogonale tRNA-Synthetase/tRNA-Paare (RS/tRNA) entwickelt, um eine ncAA an der gewünschten Stelle einzubauen, die eine breite Palette von Funktionalitäten bieten, die in ausgewählte Proteine eingebaut. Die zytoplasmatische Expression von RS/tRNA-Paaren kann ein Problem mit dem ncAA-Einbau in Wirtsproteine im Hintergrund verursachen. Die Anwendung von orthogonal übersetzenden Organellen (OTOs), die sich an das Konzept der Phasentrennung anlehnt, bietet eine Lösung für dieses Problem bei Säugetieren Zellen, was einen ortsspezifischen und proteinselektiven ncAA-Einbau ermöglicht. Bislang wurde nur Methanosarcina mazei (Mm) Pyrrolysyl-tRNA-Synthetase (PylRS) in OTOs verwendet, was das Potenzial der Methode einschränkt. Hier haben wir die Umsetzung von vier anderen weit verbreiteten orthogonalen RS/tRNA-Paaren mit OTOs untersucht, die zu unserer Überraschung nicht in der Lage waren, mRNA-selektive GCE zu erzeugen. Als nächstes testeten wir mehrere experimentellen Lösungen und entwickelten ein neues chimäres Phenylalanyl-RS/tRNA-Paar, das die ncAA Einbau in OTOs auf ortsspezifische und proteinselektive Weise. Unsere Arbeit offenbart ungeklärte Design-Zwänge beim räumlichen Engineering von Enzymfunktionen unter Verwendung von Designer-Organellen und präsentiert eine Strategie zur Überwindung dieser Probleme in vivo. Anschließend erörtern wir aktuelle Einschränkungen und künftige Richtungen der In-Cell Engineering im Allgemeinen und Protein-Engineering mit GCE im Besonderen.
Herzlichen Glückwunsch an Mikhail E. Sushkin, Marius Jung (SFB1551 IRTG-Student) und Edward Lemke!