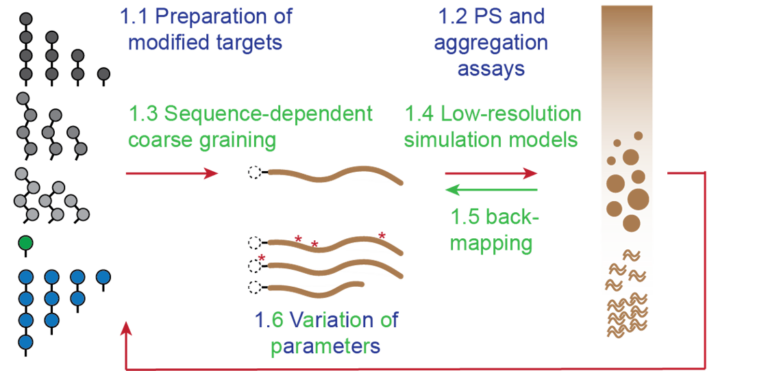
Die posttranslationalen Modifikatoren Ubiquitin und SUMO, zwei kleine, konservierte Proteine, die für ihre Fähigkeit bekannt sind, Protein-Protein-Interaktionen zu modulieren, indem sie sich kovalent an andere Proteine anlagern, zielen auf viele Proteine ab, die eine Phasentrennung (PS) durchlaufen, und wurden in die Bildung pathogener Aggregate einbezogen. Ubiquitin und SUMO wirken in erster Linie über definierte Protein-Protein-Interaktionsbereiche oder -motive. Sie modulieren jedoch auch direkt die biophysikalischen Eigenschaften ihrer Zielproteine. Mit biochemischen und biophysikalischen Ansätzen in Kombination mit molekularen Multiskalensimulationen, die auf Konzepten der Polymerphysik basieren, werden wir systematisch untersuchen, wie Ubiquitin und SUMO die Neigung von Zielproteinen, die intrinsisch ungeordnete Regionen (IDRs) enthalten, PS und Aggregation zu erfahren, beeinflussen. Anhand einer Reihe von Modell-Zielproteinen, die für neurodegenerative Erkrankungen relevant sind, werden wir den Einfluss von Ubiquitin und SUMO auf PS, Aggregation und die Eigenschaften der resultierenden Kondensate und Aggregate untersuchen. Wir werden Parameter wie den Grad der Modifikation, die Anordnung der Ubiquitin/SUMO-Einheiten, ihre Stöchiometrie und ihr Zusammenspiel mit IDRs innerhalb der Targets berücksichtigen. Parallel dazu werden wir eine Hierarchie grobkörniger Modelle entwerfen, die in der Lage sind, das Verhalten globulärer und ungeordneter Domänen sowie linearer Ubiquitin- und SUMO-Ketten genau wiederzugeben. Diese Modelle werden uns dabei helfen, den Einfluss verschiedener Merkmale der Zielmoleküle und Modifikatoren auf PS und Aggregation zu erforschen und einen Einblick auf molekularer Ebene in die entsprechenden in vitro Messungen und Anweisung weiterer Experimente durch Anpassung der relevanten Parameter. Durch das Back-Mapping dieser gröberen Modelle auf höher aufgelöste Modelle werden wir spezifische Proteinsequenzen identifizieren, die experimentell untersucht werden sollen. Längerfristig werden die Ergebnisse dieser in vitro und in silico Studien werden Vorhersagen liefern, die in zellbiologische Experimente einfließen, die die Auswirkungen von Ubiquitin und SUMO auf krankheitsrelevante Proteine untersuchen in vivo. Gleichzeitig wollen wir unseren Modellierungsansatz für die Simulation verschiedener Arten von modifizierten Proteinen verallgemeinern.