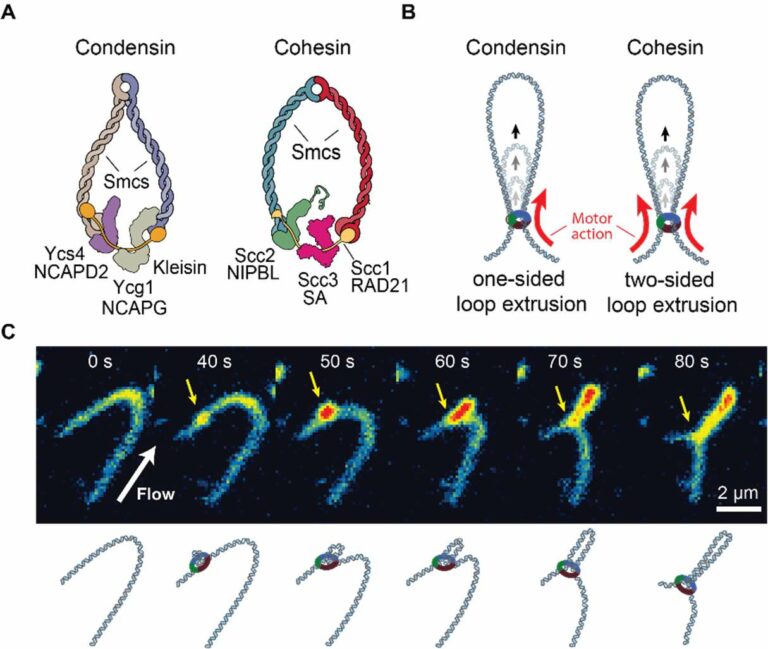
Die Verdichtung unseres zwei Meter langen Genoms in einem begrenzten, mikrometergroßen Kernvolumen stellt die Zelle vor zahlreiche Herausforderungen. Genomische Prozesse wie Replikation und Transkription führen zu dramatischen Veränderungen der DNA-Topologie, einschließlich Verschlingungen, Knoten und Verdrehungen. Diese topologisch komplexen, großen DNA-Polymere werden durch biomolekulare Kondensate gebunden, überbrückt und umgeben, darunter Nukleolus, Heterochromatin-Kompartimente und Transkriptionskondensate. Die Verdichtungsprozesse, die Chromatin zu Strukturen höherer Ordnung wie Chromatinschleifen falten, werden daher stark von diesen komplexen zellulären Umgebungen beeinflusst. Chromatinschleifen werden durch einen ATP-gesteuerten Prozess - die so genannte Schleifen-Extrusion - gebildet, der die Schleifen durch die motorische Wirkung von SMC-Komplexen (Structural Maintenance of Chromosomes) wie Condensin und Cohesin schrittweise vergrößert. Als langfristiges Ziel dieses Vorschlags wollen wir verstehen, wie der DNA-Schleifen-Extrusionsprozess mit zwei wichtigen und weit verbreiteten Phänomenen in Zellen interagiert, die in der Polymerwissenschaft gut untersucht sind: DNA-Topologie und Kernkondensate. In den ersten vier Jahren wollen wir unsere Studien auf das Zusammenspiel zwischen der DNA-Topologie und der Extrusion von Schleifen konzentrieren, indem wir Einzelmolekülexperimente und numerische Simulationen verwenden. Zunächst werden wir untersuchen, wie die Schleifenextrusion den Torsionszustand der DNA beeinflusst und wie sie mit verketteter DNA, einem bekannten Nebenprodukt der DNA-Replikation, umgeht. Wir werden beobachten, wie Kondensin- und Cohesin-vermittelte Schleifen verlaufen, wenn sie auf eine verschränkte Region treffen, und wie dies die Konformationen der verschränkten DNA verändern könnte. Wir werden unsere Studie erweitern, um herauszufinden, ob und wie die Schleifenextrusion die Dekatenationsaktivität der Topoisomerase II (Top II) moduliert. Gleichzeitig werden wir grobkörnige Simulationen durchführen, um unsere experimentellen Bemühungen zu unterstützen und die zugrundeliegenden molekularen Mechanismen aufzuklären. Als Nächstes werden wir untersuchen, wie das Vorhandensein von Knoten die Schleifenextrusion beeinflusst oder behindert, indem wir verknotete DNA präparieren und sie in Gegenwart von Kohäsin- und Kondensin-Komplexen extrudieren. Die Einzelmolekülexperimente werden von grobkörnigen DNA-Simulationen begleitet, um die relevanten Versuchsbedingungen im Voraus zu bestimmen und sie direkt mit den Experimenten vergleichen zu können. Als Nebenprodukt werden wir auch DNA-Knotenwahrscheinlichkeiten im Confinement bestimmen, die Knotendiffusion untersuchen und experimentelle Bedingungen identifizieren, unter denen Knoten sich gegenseitig durchdringen können. Die zu erwartenden Ergebnisse werden eine molekulare Grundlage für das Verständnis liefern, wie schleifenbildende SMC-Proteine gemeinsam topologisch komplexe Genome zu hoch organisierten Strukturen in Zellen falten können. Für die 12-Jahres-Perspektive planen wir, diese Studie zu erweitern, um zu verstehen, wie der Schleifen-Extrusionsprozess mit Kernkondensaten interagiert, von denen allgemein angenommen wird, dass sie DNA-Schleifen überbrücken und stabilisieren und Chromatine in funktionelle Regionen aufteilen.